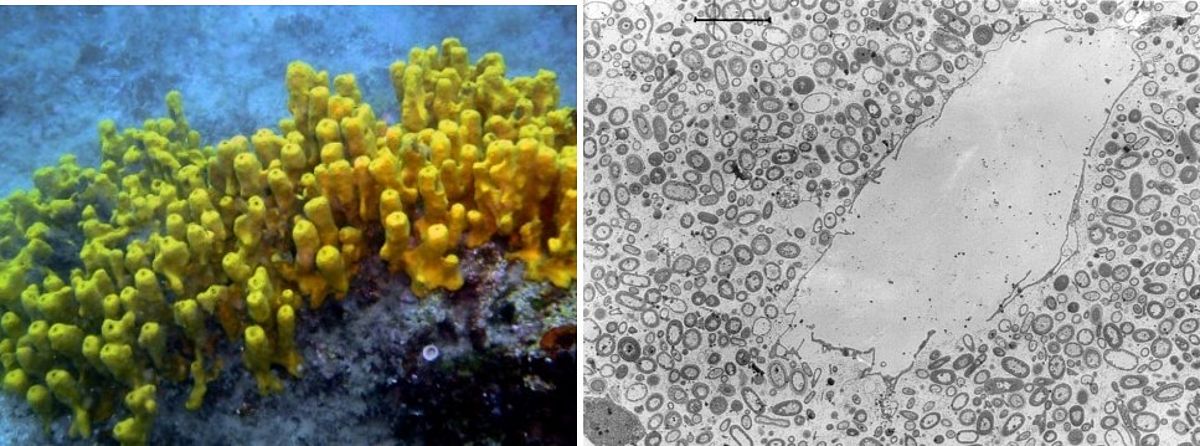
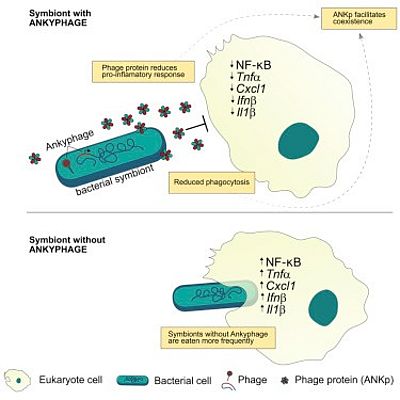
Während jüngste Studien eine große Vielfalt in der Virosphäre aufgedeckt haben, besteht die neue Herausforderung darin zu verstehen, wie Phagen die Wirt-Mikroben-Interaktionen modulieren. Wir kombinieren virale Metagenomik mit funktionellen Assays, um das Zusammenspiel zwischen Phagen, bakteriellen Symbionten und marinen Schwämmen zu untersuchen. Obwohl Schwämme massive Wassermengen durch ihre ständig arbeitenden Filterapparate verarbeiten, haben wir kürzlich gezeigt, dass sie artspezifische und sogar individuell einzigartige virale Signaturen beherbergen, die sich von anderen Umgebungen unterscheiden. Wir haben ferner ein Ankyrin-Protein (ANKp) entdeckt, das auf einem Symbiontenphagen kodiert ist und bei heterologer Expression die eukaryotische Immunantwort gegen Bakterien moduliert, wie in Makrophagen-Infektionsassays bestätigt wurde. Wir stellen die Hypothese auf, dass diese Symbiontenphagenproteine die Koexistenz im Multispezies-Wechselspiel zwischen Phagen, mikrobiellen Symbionten und Schwämmen in einer Weise erleichtern, die sich auf andere Wirt-Mikroben-Assoziationen ausdehnen kann. Interessanterweise scheinen diese "Ankyphagen" in anderen Wirts-Mikroben-Assoziationen, einschließlich des Menschen, weit verbreitet zu sein.
Wissenschaftler
Dr. Kristina Bayer, Dr. Martin T. Jahn, Dr. Laura Rix, Dr. Beate Slaby, Leon Steiner
Veröffentlichungen
Maldonado M, Bayer K, López-Acosta M (2024) Nitrogen and Phosphorus Cycling Through Marine Sponges: Physiology, Cytology, Genomics, and Ecological Implications. Frontiers in Invertebrate Physiology: A Collection of Reviews Apple Academic Press. eBook ISBN 9781003403319
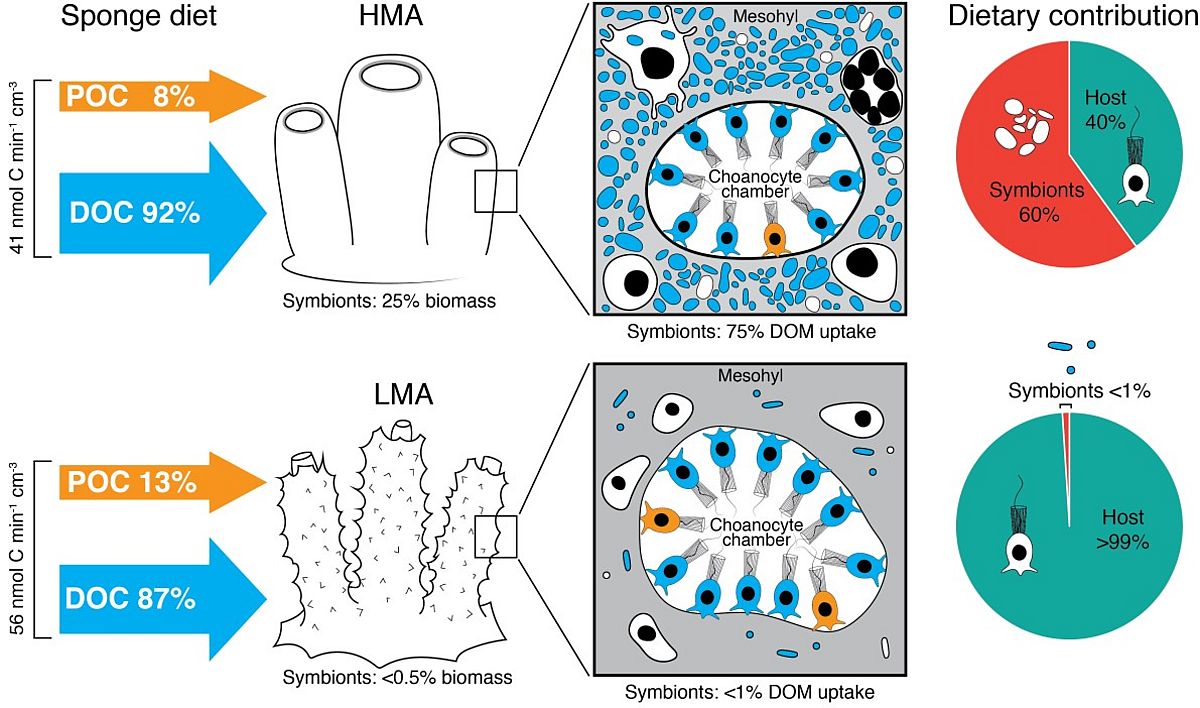
Rix L, Ribes M, Coma R, Jahn M, van Oevelen D, de Goeij J, Escrig S, Meibom A, Hentschel U (2020) Heterotrophy in the earliest gut: A single-cell view of heterotrophic carbon and nitrogen assimilation in sponge-microbe symbioses. ISME J: doi.org/10.1038/s41396-020-0706-3
Jahn MT, Arkhipova K, Markert SM, Stigloher C, Lachnit T, Pita L, Kupczok A, Ribes M, Stengel ST, Rosenstiel P, Dutilh BE, Hentschel U (2019) A phage protein aids bacterial symbionts in eukaryote immune evasion. Cell Host & Microbe 26(4):542-550.e5. doi: 10.1016/j.chom.2019.08.019.
Batani G, Bayer K, Böge J, Hentschel U, Thomas T (2019) Fluorescence in situ hybridization (FISH) and cell sorting of living bacteria. Sci Rep 9(1):18618. doi: 10.1038/s41598-019-55049-2.
Bayer K, Jahn MT, Slaby BM, Moitinho-Silva L, Hentschel U (2018) Marine sponges as Chloroflexi Hot Spots: Genomic insights and high resolution visualization of an abundant and diverse symbiotic clade. mSystems: doi.org/10.1128/mSystems.00150-18
Projektförderung
SFB 1182/2: Origin and Function of Metaorganisms
http://www.metaorganism-research.com/